Guide Ultime : Comment Utiliser l'Imputer de Scikit-learn
Updated on
Lors du travail avec de grands ensembles de données, il est presque inévitable de rencontrer des valeurs manquantes. La gestion efficace des données manquantes est une étape fondamentale dans le processus de prétraitement des données. L'Imputer de Scikit-learn offre un ensemble de stratégies robustes pour cette tâche. Dans cet article, nous plongerons dans les Imputeurs de Scikit-learn : SimpleImputer, IterativeImputer, KNNImputer, et comment les utiliser pour traiter les données numériques et catégorielles.
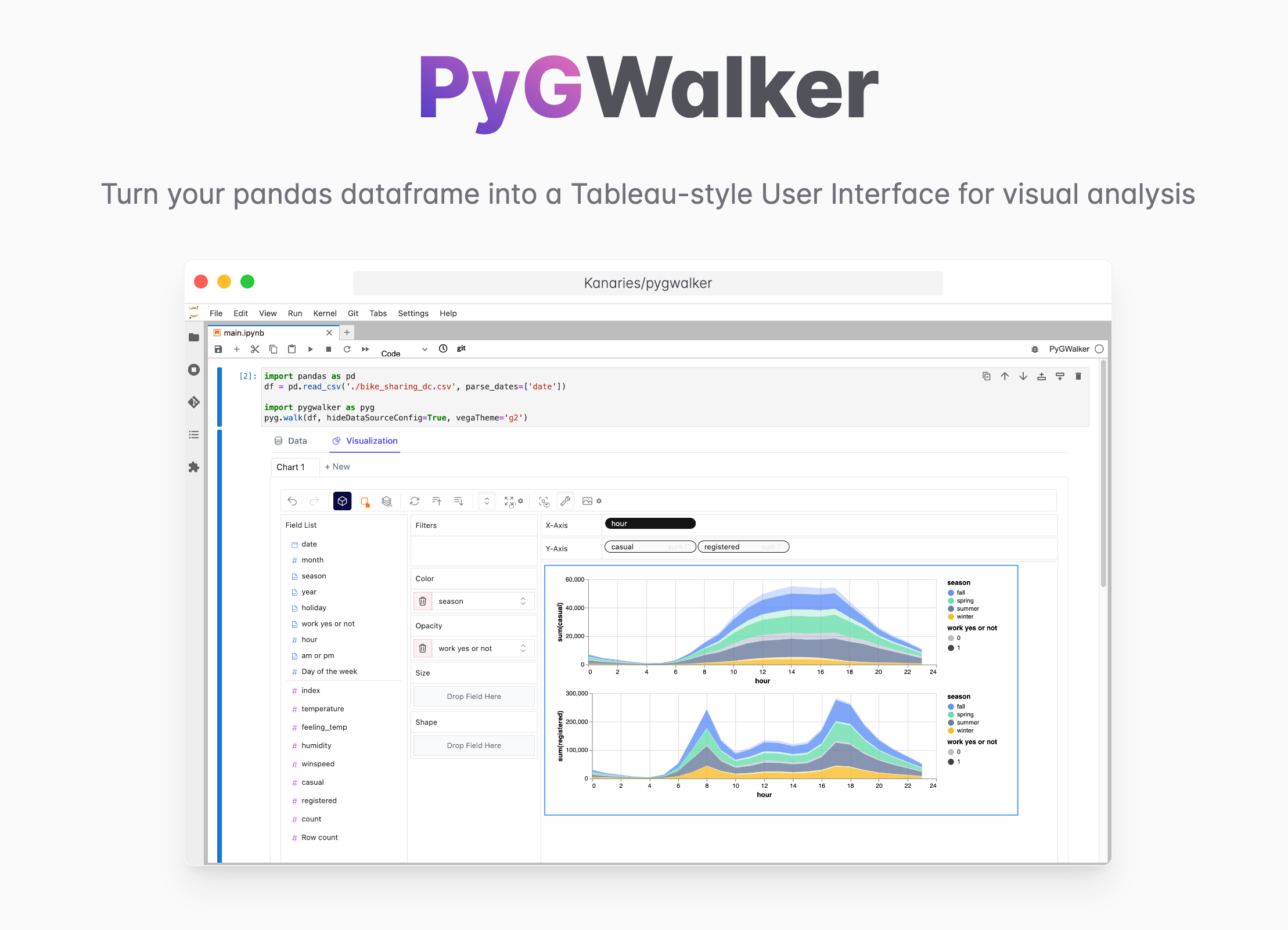
Vous souhaitez créer rapidement une visualisation de données à partir d'un dataframe Python Pandas sans code ?
PyGWalker est une bibliothèque Python pour l'analyse exploratoire des données avec visualisation. PyGWalker (opens in a new tab) peut simplifier votre flux de travail d'analyse de données et de visualisation de données dans un notebook Jupyter, en transformant votre dataframe pandas (et le dataframe polars) en une interface utilisateur de style Tableau pour l'exploration visuelle.
Démasquer l'Imputer
Avant de nous plonger dans les "comment faire", comprenons d'abord ce qu'est un Imputer. Essentiellement, un imputer est un estimateur qui remplit les valeurs manquantes dans votre ensemble de données. Pour les données numériques, il utilise des stratégies telles que la moyenne, la médiane ou une constante, tandis que pour les données catégorielles, il utilise la valeur la plus fréquente ou une constante. Vous pouvez également entraîner votre modèle pour prédire les valeurs manquantes, en fonction de la complexité de vos données et des ressources à votre disposition.
Dans ce guide, nous nous concentrerons principalement sur les Imputeurs de Scikit-learn : SimpleImputer, IterativeImputer et KNNImputer. De plus, nous vous guiderons à travers le processus de création d'un pipeline pour imputer à la fois des caractéristiques catégorielles et numériques de manière transparente et les alimenter dans un modèle d'apprentissage automatique.
Configuration de l'Imputer de Scikit-learn
Les fonctions d'imputation de Scikit-learn offrent un moyen pratique de traiter les données manquantes en utilisant quelques lignes de code. De plus, elles vous permettent de créer des pipelines, ce qui facilite la reproduction des résultats et l'amélioration de votre flux de travail de développement d'apprentissage automatique.
Environnement et ensemble de données
Avant de plonger dans les détails de l'utilisation de l'Imputer de Scikit-learn, configurons d'abord notre environnement et obtenons un ensemble de données. Pour ce tutoriel, nous utiliserons la base de données sur le diabète des Indiens Pima, un ensemble de données populaire dans la communauté de l'apprentissage automatique.
Cet ensemble de données est disponible gratuitement sur Kaggle, et nous pouvons le télécharger directement en utilisant l'API de Kaggle. Tout d'abord, nous devons cependant installer les bibliothèques Python nécessaires :
!pip install numpy pandas scikit-learn kaggleEnsuite, nous téléchargerons la base de données sur le diabète des Indiens Pima en utilisant l'API de Kaggle :
!kaggle datasets download -d uciml/pima-indians-diabetes-database
!unzip pima-indians-diabetes-database.zip -d ./datasetMaintenant que nous avons notre ensemble de données, chargeons-le dans un DataFrame pandas et jetons un coup d'œil :
import pandas as pd
df = pd.read_csv("./dataset/diabetes.csv")
print(df.head())Plongeons dans l'ensemble de données
Une fois les données chargées, il est essentiel de comprendre leur structure. La base de données sur le diabète des Indiens Pima comporte huit caractéristiques numériques et une colonne cible binaire, "Outcome". Les caractéristiques sont liées à diverses mesures de santé, telles que le niveau de glucose, la pression artérielle et l'IMC, tandis que la colonne cible indique si un individu est diabétique ou non.
Tout d'abord, examinons la forme de notre ensemble de données :
print(df.shape)Avec cette sortie, nous pouvons confirmer que notre ensemble de données contient 768 lignes et 9 colonnes.
Révéler les valeurs manquantes
Dans les ensembles de données du monde réel, les valeurs manquantes sont assez courantes. Par conséquent, avant de plonger dans la modélisation d'apprentissage automatique, nous devons identifier et manipuler de manière appropriée ces valeurs manquantes.
Vérifions s'il y a des valeurs manquantes dans notre ensemble de données :
missing_values = df.isnull().sum()
print(missing_values)Dans un scénario idéal, cette commande renverrait des zéros pour toutes les colonnes. Cependant, en pratique, c'est rarement le cas. Nous verrons comment traiter ces valeurs manquantes dans les prochaines sections.
Imputation des valeurs numériques
Dans le domaine de l'Imputer de Scikit-learn, nous distinguons l'imputation numérique de l'imputation catégorielle. L'imputation numérique est le processus de remplacement des valeurs numériques manquantes par des estimations statistiques. Il est courant d'utiliser la moyenne, la médiane ou le mode comme valeur de remplacement.
Dans le cadre de notre tutoriel, supposons que la colonne 'Pression artérielle' comporte des valeurs manquantes. Notre première tâche consiste à confirmer le nombre de valeurs manquantes :
print(df['Pression artérielle'].isnull().sum())Dans la section suivante, nous apprendrons comment imputer ces valeurs manquantes à l'aide de SimpleImputer de Scikit-learn.
Imputation des valeurs numériques avec SimpleImputer de Scikit-learn
Le SimpleImputer de Scikit-learn offre une manière simple de traiter les valeurs numériques manquantes. Il offre différentes stratégies, notamment le remplacement des valeurs manquantes par la moyenne, la médiane ou une valeur constante. Voyons un exemple d'utilisation de SimpleImputer pour imputer les valeurs manquantes dans la colonne 'Pression artérielle'.
from sklearn.impute import SimpleImputer
# Créer une instance de SimpleImputer
imputer = SimpleImputer(strategy='mean')
# Remodeler la colonne en un tableau 2D
pression_arterielle = df['Pression artérielle'].values.reshape(-1, 1)
# Imputer les valeurs manquantes
pression_arterielle_imputee = imputer.fit_transform(pression_arterielle)
# Mettre à jour le DataFrame avec les valeurs imputées
df['Pression artérielle'] = pression_arterielle_imputeeEn définissant le paramètre de stratégie sur 'mean', le SimpleImputer calcule la moyenne des valeurs disponibles et remplace les valeurs manquantes par cette valeur moyenne. Vous pouvez également utiliser 'median' ou 'constant' comme stratégie, en fonction de la nature de vos données.
Après avoir imputé les valeurs manquantes, il est toujours bon de vérifier si des valeurs manquantes subsistent :
print(df['BloodPressure'].isnull().sum())Après avoir imputé les valeurs manquantes, la sortie devrait être 0, ce qui indique qu'il n'y a plus de valeurs manquantes dans la colonne 'BloodPressure'.
Imputation des valeurs catégoriques
Maintenant, passons à l'imputation des valeurs catégoriques. Pour cet exemple, considérons la colonne 'SkinThickness', qui contient des valeurs catégoriques manquantes.
Comme pour la section précédente, nous allons d'abord vérifier combien de valeurs manquantes sont présentes dans la colonne 'SkinThickness' :
print(df['SkinThickness'].isnull().sum())Pour imputer des valeurs catégoriques, nous pouvons utiliser la stratégie 'most_frequent' fournie par le SimpleImputer. Cette stratégie remplace les valeurs manquantes par la valeur la plus fréquente dans la colonne.
# Créer une instance de SimpleImputer pour les valeurs catégoriques
imputer_categorical = SimpleImputer(strategy='most_frequent')
# Remodeler la colonne en un tableau 2D
skin_thickness = df['SkinThickness'].values.reshape(-1, 1)
# Imputer les valeurs manquantes
imputed_skin_thickness = imputer_categorical.fit_transform(skin_thickness)
# Mettre à jour le DataFrame avec les valeurs imputées
df['SkinThickness'] = imputed_skin_thicknessAprès avoir imputé les valeurs manquantes, vérifions si des valeurs manquantes subsistent dans la colonne 'SkinThickness' :
print(df['SkinThickness'].isnull().sum())La sortie devrait être 0, ce qui indique que toutes les valeurs catégoriques manquantes dans la colonne 'SkinThickness' ont été imputées avec succès.
Conclusion
Dans cette partie du guide, nous avons appris à utiliser le SimpleImputer de Scikit-learn pour gérer les valeurs manquantes dans les colonnes numériques et catégoriques. Nous avons exploré des stratégies telles que la moyenne, la médiane et la valeur la plus fréquente pour imputer les valeurs manquantes en fonction de la nature des données.
L'imputation est une étape essentielle dans le pipeline de prétraitement des données, car elle nous permet de conserver des informations précieuses et de garantir que nos modèles peuvent apprendre à partir d'ensembles de données complets. En utilisant le SimpleImputer de Scikit-learn, les scientifiques des données et les praticiens en apprentissage automatique peuvent gérer efficacement les données manquantes et construire des modèles robustes.