Pheatmap en R : Créer des cartes thermiques personnalisables
Updated on
Les cartes thermiques sont un outil essentiel dans la boîte à outils du data scientist, fournissant une représentation visuellement intuitive de jeux de données complexes. Parmi les différents packages disponibles en R pour générer des cartes thermiques, Pheatmap se distingue par sa flexibilité et ses options de personnalisation. Cet article vous guidera à travers le processus de création de cartes thermiques personnalisables et attrayantes à l'aide de Pheatmap en R.
Pheatmap est plus qu'une simple fonction en R ; c'est un outil puissant qui permet aux utilisateurs de créer des cartes thermiques avec un plus grand contrôle et des options de personnalisation que la fonction de carte thermique standard de R. Avec Pheatmap, les utilisateurs peuvent visualiser l'analyse de l'expression génique, dessiner des cartes thermiques de corrélation et personnaliser les tailles d'étiquettes et la visibilité des dendrogrammes. Plongeons dans le monde de Pheatmap et explorons ses capacités.
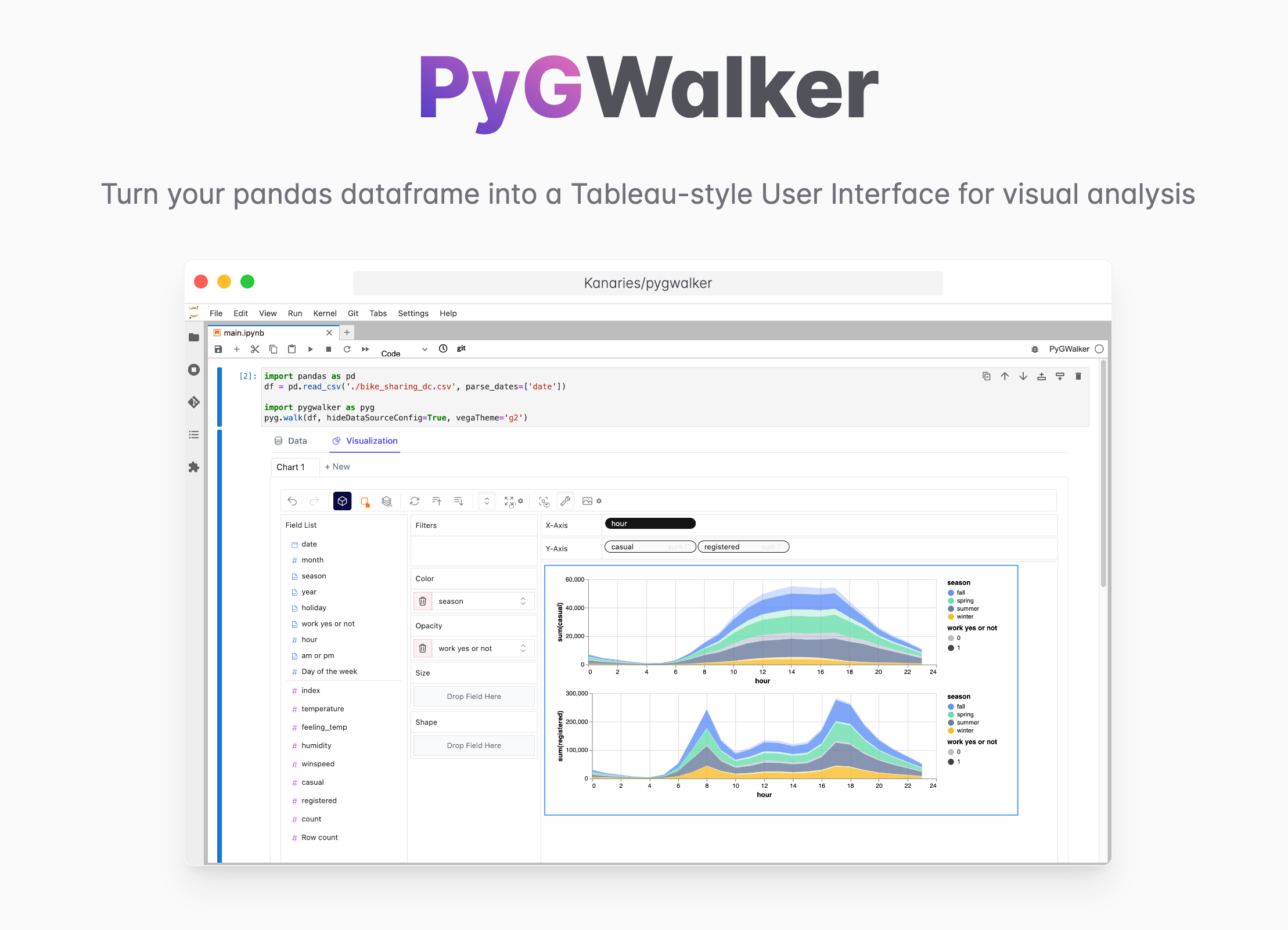
Vous voulez créer rapidement une visualisation de données à partir d'un dataframe Python Pandas sans code ?
PyGWalker est une bibliothèque Python pour l'analyse de données exploratoires avec visualisation. PyGWalker (opens in a new tab) peut simplifier votre flux de travail d'analyse de données et de visualisation de données dans les notebooks Jupyter, en transformant votre dataframe pandas (et dataframe polars) en une interface utilisateur de style Tableau pour l'exploration visuelle.
Qu'est-ce que Pheatmap en R ?
Pheatmap est une fonction en R qui génère de jolies cartes thermiques, permettant aux data scientists de visualiser des données complexes de manière simplifiée. Il offre plus de contrôle et d'options de personnalisation par rapport aux fonctions de carte thermique standard de base de R telles que heatmap() et heatmap.2(). Pheatmap se distingue par sa capacité à produire des cartes thermiques esthétiquement plaisantes et informatives.
Pheatmap est particulièrement utile en génomique, où il est souvent utilisé pour visualiser les données d'expression génique. Il permet l'ajout d'annotations et utilise des méthodes de regroupement pour regrouper des données similaires, améliorant ainsi l'interprétabilité de la carte thermique. Il offre également des options de standardisation en score Z des lignes/colonnes, qui peuvent être cruciales dans certains scénarios d'analyse de données.
Comment fonctionne Pheatmap ?
Pheatmap fonctionne en prenant une matrice de données et en la convertissant en une carte thermique visuellement intuitive. Les valeurs des données sont représentées par des couleurs dans la carte thermique, l'intensité de la couleur indiquant la magnitude de la valeur. Cela facilite l'identification des motifs et des corrélations dans les données.
La fonction effectue également un regroupement hiérarchique des données, regroupant des lignes et des colonnes similaires. Cela est représenté visuellement par un dendrogramme, un diagramme en forme d'arbre qui montre la relation hiérarchique entre les points de données. La méthode de regroupement utilisée par Pheatmap peut être personnalisée selon les besoins de l'utilisateur.
Pheatmap permet également un haut degré de personnalisation de l'apparence de la carte thermique. Les utilisateurs peuvent contrôler la palette de couleurs, les tailles d'étiquettes, la visibilité du dendrogramme, etc. Cela fait de Pheatmap un outil polyvalent pour la visualisation de données en R.
Avantages de Pheatmap par rapport à la carte thermique standard de R
Bien que la fonction de carte thermique de base de R soit utile pour la génération de cartes thermiques basiques, Pheatmap offre plusieurs avantages qui en font un choix préféré pour de nombreux data scientists.
Tout d'abord, Pheatmap offre un plus grand contrôle sur l'apparence de la carte thermique. Les utilisateurs peuvent personnaliser la palette de couleurs, ajuster les tailles d'étiquettes et contrôler la visibilité du dendrogramme. Cela permet de créer des cartes thermiques informatives et esthétiquement attrayantes.
Deuxièmement, Pheatmap effectue un regroupement hiérarchique des données, regroupant des lignes et des colonnes similaires. Cela améliore l'interprétabilité de la carte thermique et facilite l'identification des motifs dans les données.
Troisièmement, Pheatmap permet l'ajout d'annotations et l'utilisation de filtres, ce qui peut être particulièrement utile dans l'analyse d'expression génique. Il offre également des options de standardisation en score Z des lignes/colonnes, offrant plus de flexibilité dans l'analyse des données.
En conclusion, bien que la fonction de carte thermique de base de R soit un outil utile pour la génération de cartes thermiques basiques, Pheatmap offre un niveau de contrôle et de personnalisation supérieur qui en fait un outil puissant pour la visualisation de données en R.
Personnalisation de l'apparence de Pheatmap en R
L'un des principaux avantages de Pheatmap est la possibilité de personnaliser l'apparence de la carte thermique en fonction de vos besoins spécifiques. Voici comment vous pouvez le faire :
Personnalisation des couleurs
Pheatmap permet de personnaliser la palette de couleurs utilisée dans la carte thermique. Cela peut être fait à l'aide du paramètre color dans la fonction pheatmap(). Vous pouvez choisir parmi une variété de palettes de couleurs disponibles en R, ou créer la vôtre.
Personnalisation des étiquettes
La taille et l'apparence des étiquettes dans la carte thermique peuvent être ajustées à l'aide des paramètres fontsize et fontface. Cela vous permet de contrôler la lisibilité de la carte thermique et de l'ajuster en fonction de vos besoins de présentation.
Visibilité du dendrogramme
Pheatmap permet de contrôler la visibilité du dendrogramme, un diagramme en forme d'arbre qui montre la relation hiérarchique entre les points de données. Cela peut être fait à l'aide des paramètres show_rownames et show_colnames dans la fonction pheatmap().
Ajout d'annotations
Pheatmap permet d'ajouter des annotations à la carte thermique, ce qui peut être particulièrement utile dans l'analyse d'expression génique. Cela peut être fait à l'aide des paramètres annotation_row et annotation_col dans la fonction pheatmap().
En conclusion, Pheatmap offre un haut niveau de personnalisation qui vous permet de créer des heatmaps qui sont à la fois informatives et attrayantes visuellement. Que vous visualisiez des données génomiques ou dessiniez des heatmaps de corrélation, Pheatmap vous offre la flexibilité et le contrôle nécessaires pour créer de magnifiques heatmaps regroupées et personnalisables en R.
Méthode de regroupement utilisée par Pheatmap
Pheatmap utilise le regroupement hiérarchique pour regrouper les points de données similaires. Il s'agit d'une méthode d'analyse de cluster qui vise à construire une hiérarchie de clusters. Le résultat final est une représentation arborescente des données, appelée dendrogramme, qui permet aux utilisateurs de visualiser les données de manière à mettre en évidence les relations entre les points de données.
Dans Pheatmap, la méthode de regroupement peut être personnalisée en utilisant les paramètres clustering_distance_rows et clustering_distance_cols pour les lignes et les colonnes respectivement. La méthode par défaut est "euclidienne", mais d'autres méthodes comme "maximum", "manhattan", "canberra", "binaire" ou "minkowski" peuvent également être utilisées.
Tracé de heatmaps en R avec Pheatmap
Créer une heatmap avec Pheatmap en R est simple. Voici un exemple de base :
# Charger la bibliothèque pheatmap
library(pheatmap)
# Créer une matrice de données
data <- matrix(rnorm(200), 20, 10)
# Générer la heatmap
pheatmap(data)Cela générera une heatmap basique avec les paramètres par défaut. Vous pouvez personnaliser la heatmap en ajoutant des paramètres à la fonction pheatmap(). Par exemple, pour changer la palette de couleurs, vous pouvez utiliser le paramètre color :
# Définir une palette de couleurs
my_palette <- colorRampPalette(c("bleu", "blanc", "rouge"))(25)
# Générer la heatmap avec la palette de couleurs personnalisée
pheatmap(data, color = my_palette)Personnaliser les couleurs dans Pheatmap
Pheatmap permet une grande personnalisation des couleurs. Vous pouvez définir votre propre palette de couleurs et l'appliquer à la heatmap. Cela se fait en utilisant le paramètre color dans la fonction pheatmap(). Voici un exemple :
# Définir une palette de couleurs
my_palette <- colorRampPalette(c("bleu", "blanc", "rouge"))(25)
# Générer la heatmap avec la palette de couleurs personnalisée
pheatmap(data, color = my_palette)Dans cet exemple, la fonction colorRampPalette() est utilisée pour créer une palette de 25 couleurs allant du bleu au blanc au rouge. Cette palette est ensuite appliquée à la heatmap en utilisant le paramètre color.
Conclusion
En conclusion, Pheatmap est un outil puissant pour créer des heatmaps regroupées personnalisables en R. Que vous visualisiez des données génomiques, dessiniez des heatmaps de corrélation ou simplement exploriez vos données, Pheatmap vous offre la flexibilité et le contrôle dont vous avez besoin.
Questions fréquemment posées
Quels sont les avantages d'utiliser Pheatmap par rapport à la fonction de heatmap de base de R standard ?
Pheatmap offre plusieurs avantages par rapport à la fonction de heatmap de base de R standard. Il offre un plus grand contrôle sur l'apparence de la heatmap, effectue un regroupement hiérarchique des données et permet l'ajout d'annotations et l'utilisation de filtres. Cela en fait un outil puissant pour la visualisation des données en R.
Comment personnaliser la palette de couleurs dans Pheatmap ?
Vous pouvez personnaliser la palette de couleurs dans Pheatmap en utilisant le paramètre color dans la fonction pheatmap(). Vous pouvez choisir parmi une variété de palettes de couleurs disponibles dans R, ou créer la vôtre.
Quelles méthodes de regroupement utilise Pheatmap ?
Pheatmap utilise le regroupement hiérarchique pour regrouper les points de données similaires. La méthode de regroupement peut être personnalisée en utilisant les paramètres clustering_distance_rows et clustering_distance_cols. La méthode par défaut est "euclidienne", mais d'autres méthodes comme "maximum", "manhattan", "canberra", "binaire" ou "minkowski" peuvent également être utilisées.